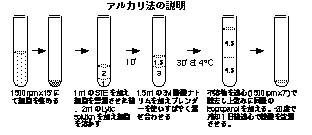
1日目,プラスミドを含んだ大腸菌を,適当な抗生物質(Amp. resistant なら 25 ug/ml Amp, Tet. resistant なら 12.5 ug/ml Tet. )を加えた培地,L broth に移植し,一日例によってスタ−ラ−で撹はんしながら培養する。
2日目,翌日,今度はSpectinomycinを含んだ等量の L brothを加え (300 ug/ml),さらに一日間培養を続ける。これによって,細胞あたりのプラスミドの数を増やす。
3日目,バクテリアを遠心(1500rpm ,20'ー30')によって集め,STE sol.にsuspend する。つぎに,この状態でスタ−ラ−で撹はんしながらLytic sol.を加える。採集的にはSTE:Lytic sol.=1:2の比となるように,ゆっくりと加える。
| STE sol. | ショ糖 | 0.25M | |
| トリス塩酸 | 50 mM (pH 8.8) | ||
| EDTA | 50 mM | ||
| Lytic sol. | NaOH | 0.2N | |
| SDS | 1 % | ||
通常は,低温下で,リゾチ−ム(0.15M NaCl, 0.1M EDTA, pH 8.0 )を加え,細菌細胞を溶かしてから,上記のLytic solutionを加えるが,簡便のため,Chroloformで代用している。Chroloform は,細菌細胞を溶かす作用がある。
あらかじめ,Lytic sol. 20 ml をスタ−ラ−で撹はんしているところに,バクテリアのペレットをSTE sol. 10 ml で suspendしながら加えていく。すると,みるみるうちに溶液は粘調になっていく。DNAが溶け出してくるためである。
余談だが,SDSは,K+ イオンの添加で,カリウムSDS複合体を形成し,難溶物となる。したがって,一般に,SDS沈澱を行う以外に,SDSを含む溶液にカリウムを使ってはいけない。
十分に溶けた(30' 以上)ところで,3M酢酸ナトリウム(pH 4.8)を,溶菌液:酢酸ナトリウム=30:15の比でゆっくりと加える。すると,pHがアルカリから産生に変わり,タンパク質等が沈澱する。その際,通常のDNAは急激なpHの変化のためannealすることができず不溶性となり,沈澱の中に混じってしまう。
この状態で,4゚C に30' 以上静置する。その後,遠心を行ない上澄みを回収する。ガ−ゼなどを用いてできるだけ沈澱物を混入させないように気をつける。この上澄みのなかに,急激なpHの変化にもめげず,annealして可溶性のまま残ったDNA(circular DNA, hairpin DNA など)が含まれている。

核酸の沈澱はエタノ−ルでもよいが,イソプロパノ−ルのほうが混ぜる量が半分で済むので,それだけ核酸の希釈が起こらず回収しやすくなる。イソプロパノ−ルは,1:1の割合で加える。(ちなみにエタノ−ルは1:2)
上澄み1に対して上記の割合で,冷したIsopropanol あるいは, EtOH を加える。すると,溶けていたDNAやRNA,多糖などが,沈澱してくる。本来ならこれを−70゚Cで冷せればいいのだが,それもままならない場合は,−20゚Cでも時間を多くかければ大丈夫である。
十分に冷却した後,これを遠心にかける(できるなら超遠心が望ましいが,低速でも時間を多くとれば大丈夫;多少沈澱しないのもあるが,気にしない気にしない・・・)。
沈澱には,目的のプラスミドDNAの他にRNAが大量に含まれている。また,タンパク質,多糖類もかなりまだ混入している。そして,酢酸も残っているので,pHもまだ,かなり低い。そこで,この後は,通常のSDS−フェノ−ル処理を行なってタンパク質,多糖類などのコンタミを除去するとともに,場合によってはSDS−フェノ−ル処理の前,あるいは2度目の処理の前にRNase A, T1 などで過剰のRNAを分解する。ただし,DNAの量が少ない場合は,EtOH沈澱をする際のキャリア−となるので,ある程度はRNAが入っていたほうがよいようである。 また,SDS−フェノ−ル処理を行なった後は,必ず,クロロホルムによる混合,遠心を行ない,フェノ−ルを除去する。
この方法はバクテリアに限らず,細胞内に環状の核酸,あるいはヘアピン状の一本鎖分子を含んでいる生物についても利用できる。